在学习GWAS,求Q矩阵。试了一下STRUCTURE,报错一直没解决,不知道为啥。(Windows的没试,用的Linux虚拟机)
所以换ADMIXTURE试试看先。
ADMIXTURE
功能: ADMIXTURE 类似于 STRUCTURE,但具有更快的计算速度,适合大规模数据集。
特点: 它使用最大似然估计方法来估计群体归属和个体的混合比例。
适用范围: 适合处理大规模 SNP 数据,计算效率高。
官网:ADMIXTURE
1.下载
进入官网,选择合适的版本进行下载

如果你是Mac就下载下面的,好像没有Windows版本的。
(2)解压
建一个新的文件夹,把下载下来的安装包拖到新的文件夹中进行解压
tar -zxvf admixture_linux-1.3.0.tar.gz
解压完了会有这些文件

(3)添加环境变量
sudo vim ~/.bashrc
export PATH=$PATH:/home/cb2/admixture/dist/admixture_linux-1.3.0
source ~/.bashrc
这里vim编辑器的使用不介绍了,把:后面的改成自己的路径即可,不知道的话就pwd一下
后面是重新加载一下文件
(4)检查是否安装成功
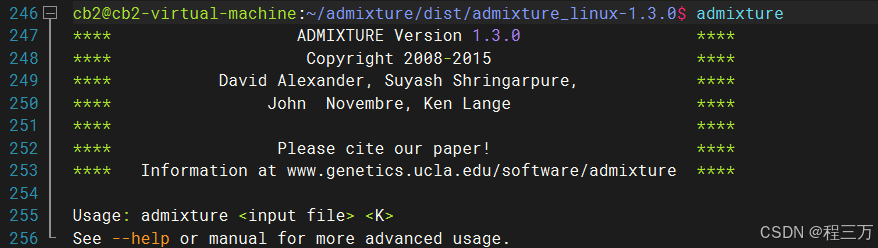
安装成功的话会有以下提醒
2.输入文件格式
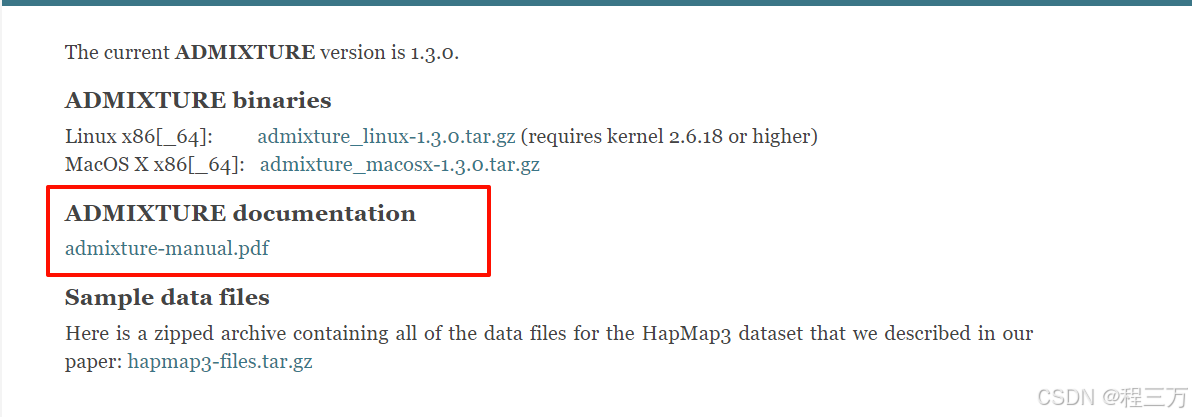
最好看一下这个pdf文件
数据如果是plink的bed文件,那还要包含bim和fam文件
数据如果是plink的ped文件,那还要包含map文件
3.K值
K值根据实际情况进行设置,通过比较得到最佳K值,admixture使用交叉验证确定K值
for K in {1..10}; do ./admixture –cv=10 1G_new.bed $K | tee log${K}.out; done
把这里的1G_new.bed换成你自己的文件名就可以了
跑这个程序需要一点时间的,耐心等待一下就好
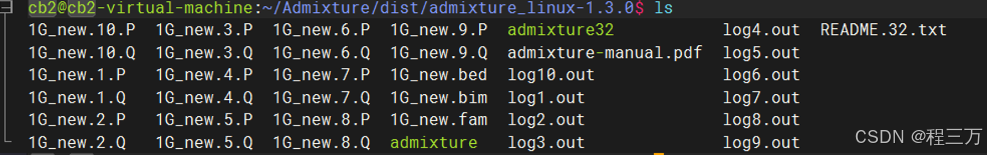
程序跑完会生成很多新的文件,每个K值都会生成两个文件,.P和.Q
.P:储存推断的祖先种群的等位基因频率
.Q:每个样本中各个祖先种群所占的百分比
然后我们选择CV值最低的K值为我们的分群数目
grep -h CV log*.out
 网硕互联帮助中心
网硕互联帮助中心



评论前必须登录!
注册